Introducción
El cáncer de pulmón no microcítico sigue siendo el tipo más frecuente y primera causa de muerte por cáncer, especialmente entre el genero masculino.
Se van identificando, cada vez, más genes y alteraciones genéticas que contribuyen al desarrollo de este tipo de cáncer, por lo que a su vez, se van desarrollando tratamientos cada vez más específicos frente a esas alteraciones genéticas.
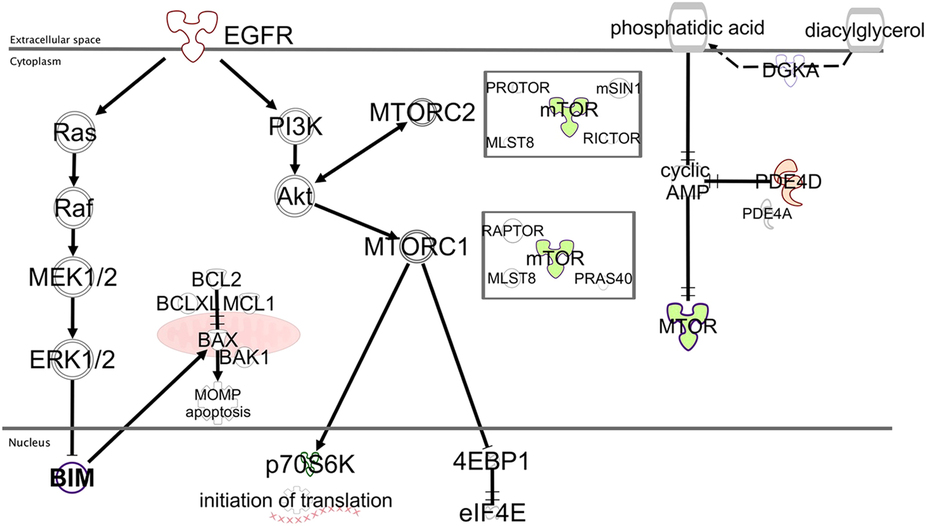
Una de las alteraciones más importantes detectadas en el cáncer de pulmón, es la mutación del Receptor del Factor de Crecimiento Epidérmico (EGFR en sus siglas en inglés), una proteína que interviene en la proliferación celular necesaria para la regeneración, en este caso, de la mucosa pulmonar.
Esta mutación lo que hace es sobre-expresar la actividad de EGFR, por lo que aumenta de forma hiperactiva la proliferación celular, y en vez de regenerar la mucosa, lo que se provoca es un tumor descontrolado.
Como hemos dicho, a medida que se van identificando alteraciones genéticas, se van desarrollando fármacos para controlarlas. En este caso los fármacos desarrollados son los de la familia de los Inhibidores de la Tirosina Quinasa (TKI en sus siglas en inglés), que impiden la hiperactividad del EGFR y por consiguiente, frenan la proliferación celular que conduce al desarrollo del tumor.
En esencia, si en un tumor, identificamos la alteración de EGFR podremos tratarlo con Inhibidores de Tirosina Quinasa, con cierta garantía de éxito. ¡Bueno!, esto en un mundo ideal ya que en el mundo real, el tumor se intenta proteger, y o bien tiene resistencias de inicio, o bien si no las tenía, las adquirirá, por lo que el tratamiento, tendrá su efectividad comprometida con el tiempo
¿Existe alguna manera de identificar que tumores van a ser más sensibles a estos TKI’s?
Bueno, pues en esas estamos. El intento, es el de identificar por medios moleculares algún marcador que nos prediga si la respuesta al tratamiento será buena o no, ya sea en una biopsia del tumor, en células tumorales circulantes en sangre, o directamente en hélices de ADN tumoral que circulan por el torrente sanguíneo. El caso es identificar moléculas, genes o mutaciones, que nos ayuden a predecir la respuesta.
En la revista Nature, este 7 de diciembre, se ha publicado un artículo de Niki Karachaliou (BIM and mTOR expression levels predict outcome to erlotinib in EGFR-mutant non-small-cell lung cancer) que arroja algo de luz sobre este tema. El trabajo ha sido dirigido por el Dr. Rafael Rosell, eminente oncólogo muy especializado en la investigación del Cáncer de Pulmón.
Los investigadores del Instituto Oncológico Dr. Rosell han identificado dos proteínas que pueden predecir los resultados del TKI, Erlotinib (Tarceva®). Estas proteínas son: BIM, una proteína que actúa en el inicio de la apoptosis o suicidio celular desencadenado por los inhibidores de la tirosina quinasa y el mTOR, que por su parte regula negativamente esa apoptosis, impidiendo la muerte celular.
El estudio encontró que los pacientes tratados con erlotinib y que poseían un alto nivel de BIM tenían menor riego de que su enfermedad progresase tras el tratamiento y por consiguiente, tuvieron un menor riesgo de mortalidad con respecto a aquellos pacientes con menores niveles de esta proteína.
En lo que respecta a mTOR, tras analizarla en aquellos pacientes con alto nivel de BIM, vieron que si sus niveles eran altos, la enfermedad progresaba más y su superviviencia era menor.
Conclusiones
Pese a que el estudio es pequeño y retrospectivo, sus conclusiones abren la posibilidad de contemplar nuevos marcadores para predecir la respuesta a estos tratamientos tan específicos.
Por otra parte, el estudio también puede sugerir que la aplicación conjunta de inhibidores de tirosina quinasa con inhibidores de mTOR (otra fámilia de fármacos) podría llegar a ser útil. Pero esa, ya es otra historia.
Estudios de GIDO en la identificación de indicadores de respuesta a Inhibidores de Tirosina Quinasa
En esta dirección, diferentes hospitales del grupo GIDO están realizando un trabajo para la identificación de un perfil metabolómico* que permita identificar qué pacientes van a responder a los inhibidores de tirosina quinasa y cuales no.
Las rutas metabólicas en el cáncer sufren una reprogramación, que produce metabólitos diferentes a los de las células normales. La identificación de éstos, podría ser la expresión de las distintas poblaciones celulares, y así poder identificar aquellos pacientes resistentes a la medicación.
Una de las ventajas de este procedimiento es que no precisa tejido tumoral, ya que el análisis se realiza en sangre.
Referencia
Niki Karachaliou, Jordi Codony-Servat[…]Rafael Rosell. BIM and mTOR expression levels predict outcome to erlotinib in EGFR-mutant non-small-cell lung cancer. Nature. Scientific Reports 5, Article number: 17499 (2015) doi:10.1038/srep17499
Definiciones
*La metabolómica es el estudio científico de los procesos químicos que involucran metabolitos. Específicamente, se podría definir como el “estudio sistemático de las huellas únicas que dejan los procesos celulares específicos en su paso”, es decir, el estudio del perfil de los metabolitos de una molécula pequeña. (Wikipedia https://es.wikipedia.org/wiki/Metabol%C3%B3mica)